基于嵌合DNA探针辅助连接酶链式反应的电化学传感策略
RNA单碱基突变在人类疾病中发挥着重要作用。 因此,准确、灵敏的RNA单碱基突变检测在一些疾病的诊断中发挥着重要作用。 然而,在临床样本中,由于大量野生型RNA中通常存在微量RNA单碱基突变,目前临床常用的qRT-PCR等RNA检测方法在临床样本的检测中遇到了挑战。
近日,福建医科大学林新华教授课题组与福建医科大学附属第一医院陈金元博士等合作,构建了基于嵌合DNA探针辅助连接酶链的电化学传感策略(cmDNA-eLCR)反应。 用于检测RNA中的单碱基突变。 本研究首先设计了3'端修饰有两个核糖核苷酸的短链DNA探针(嵌合DNA探针,S-cmDNA)。 在T4 RNA连接酶2的作用下,可以将目标RNA作为模板。 互补的cmDNA探针与正常DNA探针连接形成长的cmDNA探针(L-cmDNA)。 然后以该L-cmDNA探针为模板,在DNA连接酶的作用下,进行连接酶链式反应(chain,LCR),产生大量双链带有巯基和生物素的双链DNA产物。结束。 最后,生成的双链DNA产物通过Au-S键固定在BSA修饰的金电极表面。 当滴加SA-时,HRP上的亲和素与DNA双链产物的生物素结合,从而固定HRP。 金电极表面,催化TMB底液产生电化学电流信号,进而定量检测目标RNA(如图1所示)。
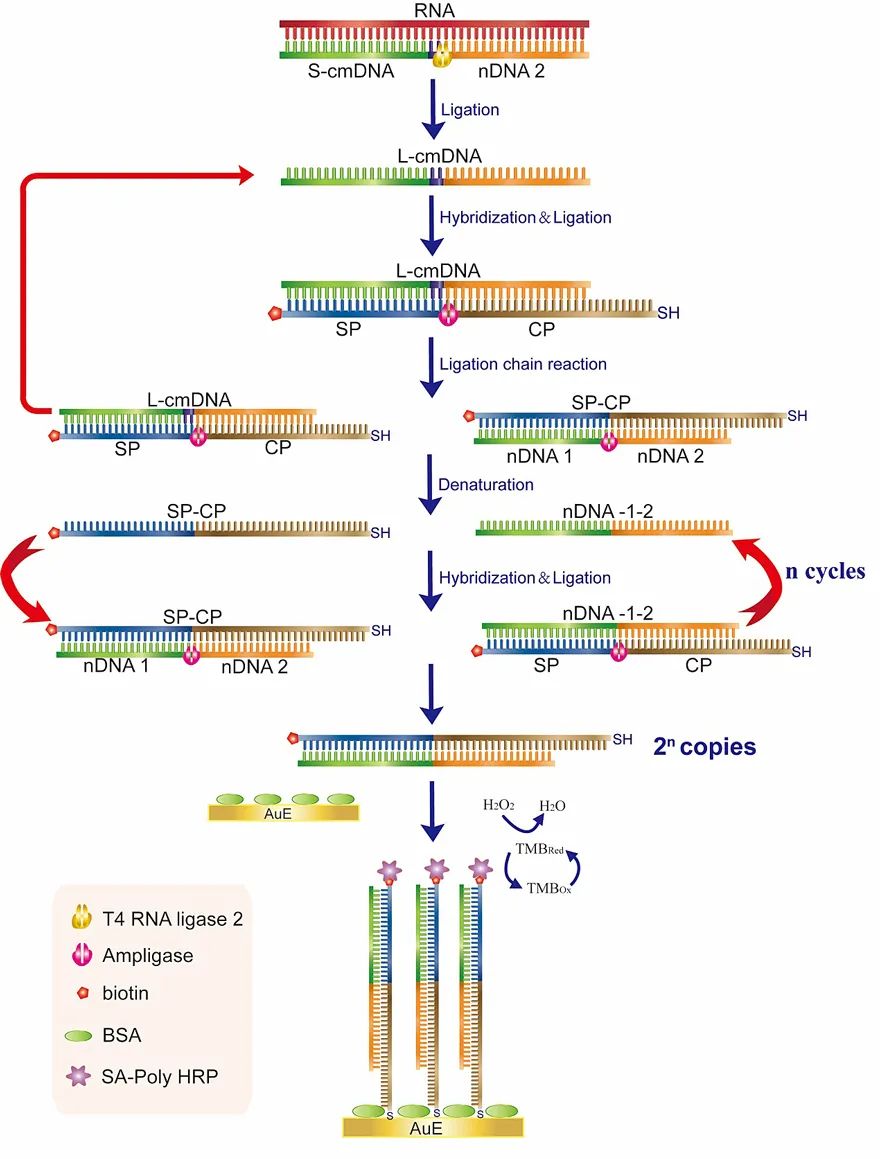
图1.基于嵌合DNA探针辅助连接酶链式反应电化学传感技术检测RNA单碱基突变示意图。 图片来源:肛门。 化学。
本研究结合了 FAM 荧光素标记的 DNA 探针和变性聚丙烯酰胺凝胶电泳方法进行表征。 结果表明,只有S-cmDNA才能产生有效的连接,并且在连接位点的碱基上具有较高的特异性(如图2A所示)。 此外,图2B的结果表明第一步的反应混合物不会干扰LCR反应,并且连接产生的L-cmDNA可以启动LCR,因此该方法不需要额外的分离步骤。 研究结果表明构建的cmDNA-eLCR具有优异的灵敏度。 在1.0×10-15 M和1.0×10-10M浓度之间,电流强度的对数与目标RNA浓度呈现良好的线性关系,检测限为0.6 fM(图3)。 即使在检测靶点浓度较高的情况下,该方法仍具有较高的特异性,能够识别溶液中的单碱基突变(如图4所示),为检测含有大量野生突变的复杂临床样本提供了重要信息。系列。 确保。 另外,在1ul NB4细胞RNA提取液和低浓度目标RNA存在的情况下,该方法表现出较强的抗干扰能力,能够准确检测溶液中微量的目标RNA,表明该方法优于已知方法。在以突变RNA为标志的相关疾病的诊断中具有广阔的应用前景。
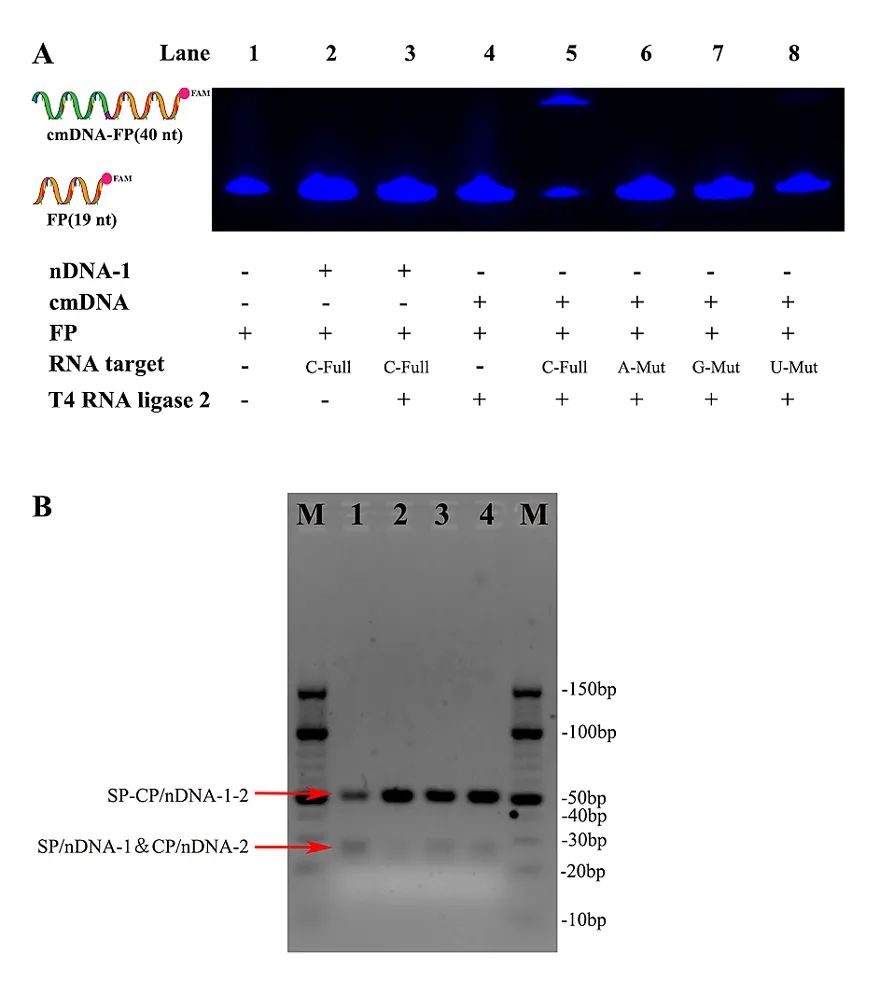
图 2. (A) 含有不同组分的第一步连接反应产物的 15% 变性聚丙烯酰胺凝胶电泳表征。 (B) 第一步反应体系对下游LCR扩增的干扰考察(以3.5%琼脂糖凝胶电泳表征):泳道1为不含目的cDNA的LCR系统产物,泳道2为LCR系统产物含有目标 cDNA。 泳道3为含有目标RNA的cmDNA-LCR体系的产物,泳道4为将第一步反应混合物(不包括RNA)添加至正常LCR体系(含有目标cDNA)后的扩增产物。 图片来源:肛门。 化学。

图3.在最佳实验条件下,所构建的cmDNA-eLCR方法检测不同浓度RNA的it曲线(A)和浓度曲线(B)。 图片来源:肛门。 化学。
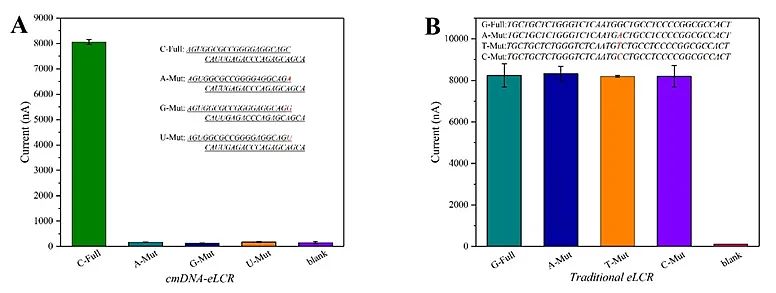
图 4. (A) 构建的 cmDNA-eLCR 在高靶浓度 (100 pM) 条件下检测完全互补的靶 RNA 和单碱基错配 RNA。 (B) 在相同的实验条件下,使用传统的eLCR检测高浓度的完全互补cDNA和单碱基错配cDNA。 图片来源:肛门。 化学。
该结果最近发表在该杂志上。 文章通讯作者为福建医科大学林新华教授和福建医科大学附属第一医院陈金元医生。 第一作者为福建医科大学附属第一医院钟光贤医生。
原文(扫描或长按二维码识别后直接进入原文页面):

通过 DNA 探针辅助链分析带碱基的 RNA
钟光贤、叶辰六、魏红祥、杨良勇、魏青霞、刘周杰、付冷西、林新华*、陈金元*
肛门。 化学,2021, 93, 911–919, DOI: 10.1021/acs..
导师介绍
林新华

本文版权归X-MOL(x-mol.com)所有。 未经许可严禁转载! 欢迎读者朋友朋友圈或微博分享!
部分文章来自互联网,如有侵权请联系删除。发布者:28预订网,转转请注明出处:https://www.28368.cn/fenxi/17321.html

